Om metoden
Som et indviklet puslespil
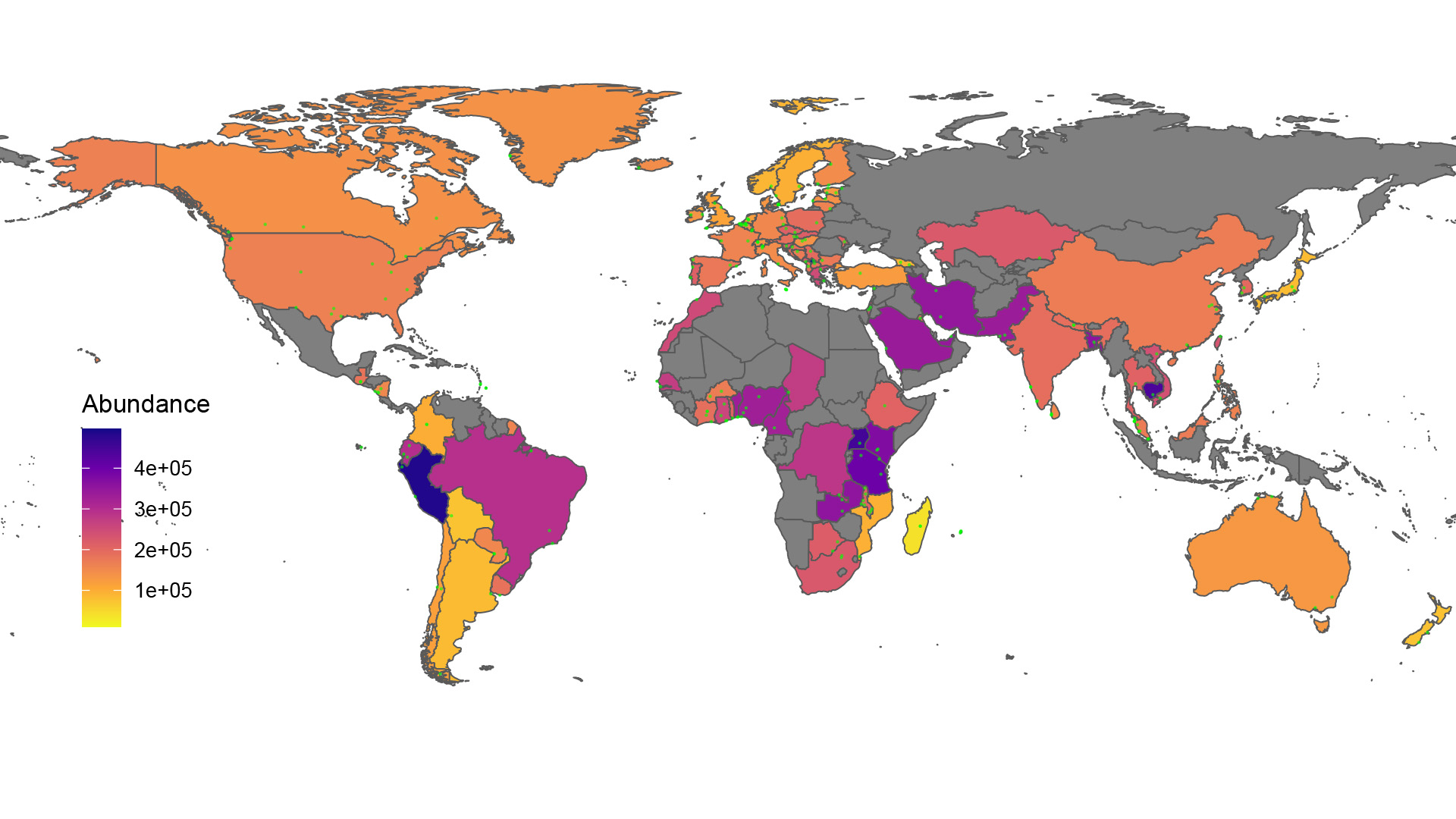
Forskningsgruppen for Genetisk Epidemiologi på DTU Fødevareinstituttet har udviklet og vedligeholder en af verdens mest omfattende resistensdatabaser. Den omfatter på nuværende tidspunkt 3134 kendte resistensgener. Den har forskerne brugt i det nye studie til at kortlægge resistensgener i spildevandsprøverne.
I prøverne findes et meget stort antal mikroorganismer, som blandt andet kommer fra menneskers afføring. De frosne spildevandsprøver er blevet sendt til DTU, hvor laboranter hiver alle bakterierne ud af de optøede prøver.
Bakterierne bliver derefter slået i stykker og deres kollektive DNA slås i mindre stykker, som state-of-the-art DNA-sekvenseringsudstyr kan læse på én gang.
De milliarder af læste DNA-sekvenser kan en supercomputer så sammenligne med kendte gener og bygge sammen til større stykker af de originale genomer, der var i prøverne. Denne proces giver bl.a. indsigt i, hvilke bakterier og genetiske nabolag resistensgenerne befinder sig i.